NAT NANO丨基于DNA测序读数的纳米尺度膜蛋白空间组织分析
作者:秦振恒
细胞通过细胞表面被称为膜受体的特殊蛋白质感知细胞外信号,如蛋白质配体。膜蛋白的纳米环境,即围绕膜受体的蛋白的组成和空间组织是动态的,经常受到配体结合的调节,这表明它们具有功能相关性。在质膜上的蛋白大多数不是均匀分布的,而是纳米尺度的动态分布。研究他们的功能相关性,需要能够全面分析细胞群中膜蛋白纳米结构的组成和空间组织的方法。
近日来自于瑞典卡罗林斯卡医学院的Ana Teixeira课题组在国际学术期刊《自然-纳米技术》(Nature Nanotechnology.)上发表重要论文《A DNA-nanoassembly-based approach to map membrane protein nanoenvironments》描述了一种非显微镜为基础的膜蛋白纳米结构域集成分析方法。
这种被称为膜纳米结构域纳米尺度解译(NanoDeep)的方法是基于DNA纳米组装将膜蛋白纳米结构信息转化为DNA测序读出。利用NanoDeep,作者对与癌症相关的Her2膜受体的纳米环境进行了表征。重要的是,NanoDeep可以通过设计用于分析的蛋白质库进行调节。NanoDeep有潜力为了解膜蛋白纳米环境的组成和空间组织调节动能提供新的见解。
NanoDeep是一种将膜蛋白空间组织信息转换为DNA测序读数的方法(图.1)。该方法通过设计一个DNA纳米组件,将其命名为纳米梳(NanoComb),该组件由四个周期性的从DNA双链骨架行伸出单链DNA探针组成。NanoComb探针包含一个条形码序列,用来标识位置。第一个探针被定义参考链,其余的是探测链。参考链预装载亲和物能识别参考蛋白(Her2蛋白)(图.1a)。带有参考蛋白结合物的NanoComb与固定的细胞孵育后,添加了一个待分析的亲和体文库(图.1b)。亲和物的寡链核苷酸包含和NanoComb检测链互补序列,为了防止未与膜蛋白结合的亲和物与NanoComb检测链结合,在加入之间使用互补链对亲合体ssDNA链进行链封闭,然后洗掉未结合膜蛋白的文库,再加入入侵连置换出亲合体的单链(图.1c)。随后通过聚合酶将NanoComb探针和亲和体DNA杂交链补齐,最后加入限制性内切酶得到既含有位置条形码又含有亲和物条形码的dsDNA(图1.d-f)。通过下一代测序分析,得到细胞群体中参考蛋白周围纳米环境的空间组织和有关组成(图.1g)。
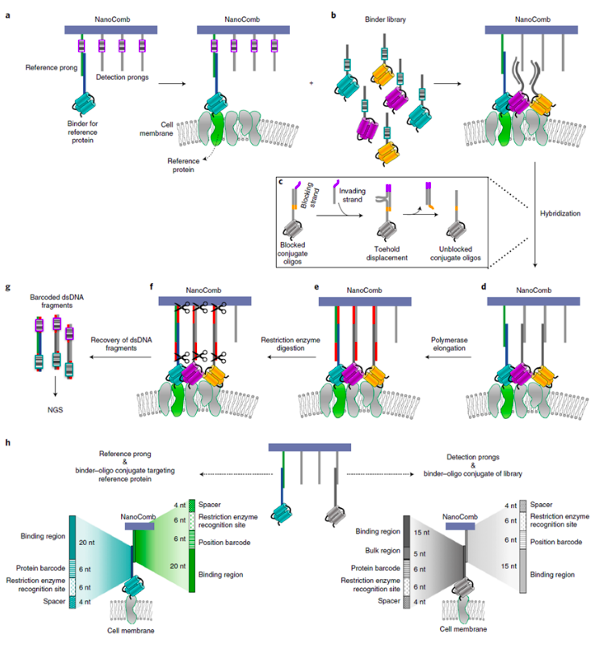
图1. (a) 包含四条单链的NanoComb通过预载参考蛋白特异性寡核苷酸亲合体,并与固定细胞培养。(b)加入融合DNA短链的亲合体文库,亲合体短链ssDNA用互补链进行封闭保护,洗去未与膜蛋白结合的亲合体。(c,d)加入入侵连对封闭链进行置换后和NanoComb探针杂交结合。(e,f)通过随后的聚合反应和补齐杂交链的单链部分,最后进行限制性酶切。(g)产生一端带有NanoComb位置信息条形码,另外一端带有亲合物条形码的dsDNA短链,PCR扩增后进行下一代测序分析(NGS)。(h)NanoComb探针和亲合体杂交的ssDNA链结构。
NanoComb是有一个100nt长的ssDNA骨架和四个较短的ssDNA退火而成(图.2a,b)。四条段的ssDNA与骨架ssDNA是周期性互补的所以伸出的四条ssDNA探针是同方向的。表面等离子共振实验和非变性PAGE电泳实验表明带有四条探针NanoComb生成(图.2c,d)。选用小而且亲和力高的亲合体作为结合物,是为了减少结合物本身大小产生的影响。为了检测亲和物与NanoComb杂交结合,使用表面等离子共振实验(SPR)分别测定了正向和反向抗Her2亲合体寡链结合物和NanoComb的结合(图.2e,f)。表明NanoComb和亲和物能够特异性的结合。
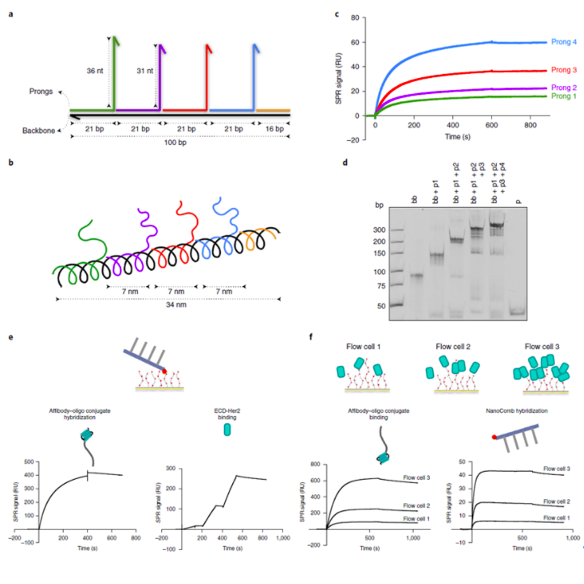
图2. (a,b)NanoComb组装结构示意图。(c,d)SPR和非变性PAGE电泳实验表明带有四条探针的NanoComb形成。(e,f)SPR 实验表面分别孵育NanoComb和参考蛋白,进行正向和反向检测NanoComb能够与亲合体结合。
在以上NanoDeep的方法流程中,目标识别步骤:包含预装载NanoComb的抗Her2亲和物和Her2的结合。检测步骤:将包含抗Her2,抗Her3和抗EGFR亲和物(结合文库)结合到它们的膜Her2和Her3上,随后结合到NanoComb上的检测链上。亲合物只有在结合目的膜蛋白后才与NanoComb检测链杂交是至关重要的。为此作者开发了一种基于立足点介导的链置换策略。为了验证DNA亲和物在保护链被置换后能与Her2 NanoComb上的检测链杂交,分别使用了Her细胞外结构域(ECD-Her2)和Her3细胞外结构域(ECD-Her3)固定于表明进行SPR分析(图.3a)。随后加入结合NanoComb的抗Her2亲和物孵育洗脱后,加入带有封闭链保护的抗Her3的寡链结合物,然后加入入侵连1,抗Her3寡链结合物与NanoComb检测链结合,接着加入入侵连2其能够使抗Her2亲和物与参考链分开。SPR分析表明只有加入入侵连1使抗Her3寡链结合物与NanoComb结合才能在SPR表面检测到NanoComb存在(图.3a左)。相反没有加入入侵连1的SPR信号显著下降(图.3a右)。
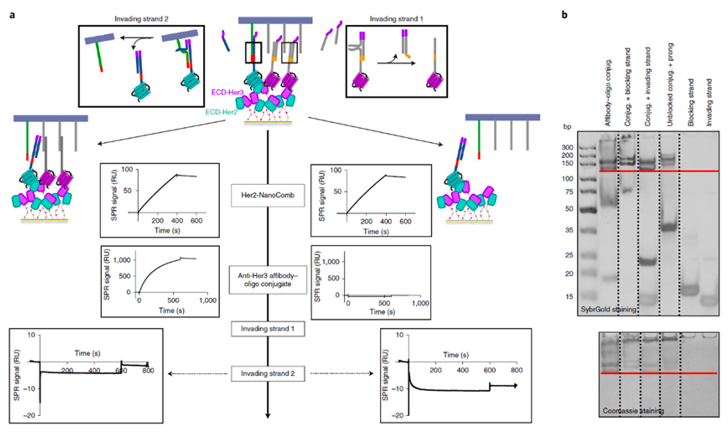
图3. (a)基于立足点介导的链置换策略,左测加入侵连1抗Her3亲和物能够与NanoComb结合,随后加入入侵连2打开和参考链杂交的抗Her2亲和物,对于没有与Her3亲和物的NanoComb就会解离,SPR信号相应就会降低,(b)非变性的PAGE电泳实验同样表明了在入侵连存在的链置换能够使亲和物和NanoComb之间的结合。
一旦NanoComb和寡链结合物结合随后需要两步酶促反应。使用SPR测定经过酶处理后NanoComb从表面脱落(图.4ab)酶促反应仅在Flow cell 1上进行,其SPR信号明显下降。利用非变性PAGE电泳进一步证实了只有酶促反应后的产物为模板PCR才能看到生成的dsDNA。
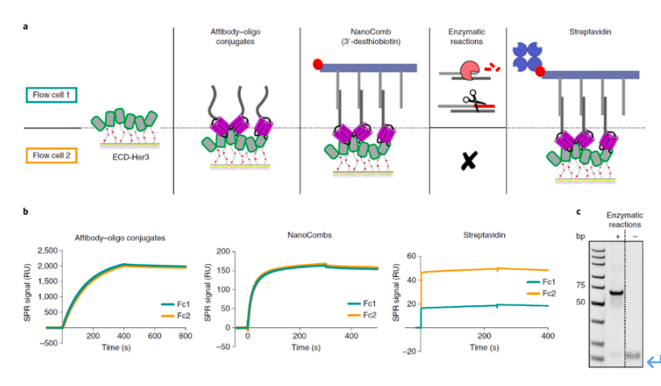
图4. (a,b)SPR实验分别检测Flow cell 1进行了酶促反应,Flow cell 2未进行酶促反应。经过酶促反应NanoComb骨架链解离导致SPR信号降低。(c)PAGE电泳实验表明只有经过酶促反应的NanoDeep实验才能检测到PCR扩增产物。
为了验证NGS分析是否能够反应表面蛋白的密度信息。我们在SPR表面进NanoDeep测试,在SPR表面结合和不同的浓度的ECD-Her2和ECD-Her(图.5a左图).经NanoDeep测试我们得到了两种浓度下的NSG分析信号,将其与SPR信号进行对比,NSG和SPR都能正确显示两种不通浓度。接下来创建ECD-Her2、ECD-Her3和ECD-EGFR不同组合的表面SPR分析以及进行NanoDeep测试,SPR和NSG信号分析每种结合文库都对其各自的靶标具有特异性,并且检测探针能特异性的检测表面存在的蛋白。

图.5 (a)1:1混合的ECD-Her2 and ECD-Her3孵育到SPR表面上后加入Her2-NanoCombs 和包含 anti-Her2 and anti-Her3的结合文库进行NanoDeep分析,NGS结果和SPR结果成正相关。(b)创建ECD-Her2、ECD-Her3和ECD-EGFR不同组合进行NanoDeep测试。
在活细胞中进行NanoDeep测定。我们使用NanoDeep分析Her2在SKBR3和MCF7乳腺癌细胞中的纳米环境,这两种细胞分别表现出Her2的高表达水平和低表达水平。对于Her2高表达的SKBR3表型用不同浓度的Her2 NanoComb处理,NGS分析结果和Her2 NanoComb浓度呈线性(图6.a)。利用NanoComb检测两种细胞株中Her2的表达是不同的(图6b)。为了检测NanoDeep是否能够检测配体对膜蛋白表达量的影响,使用HRG-β1(调节生长因子同系物)对饥饿处理的SKBR3细胞进行处理发现配体处理后的细胞Her2和Her3占有率同时增加。
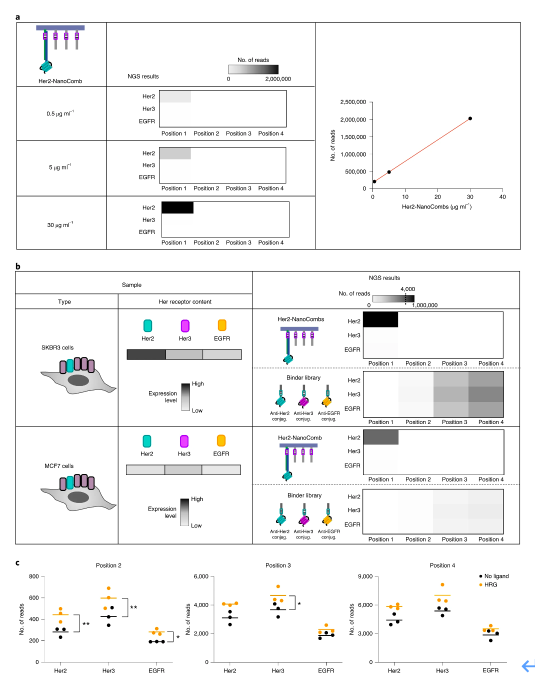
图6.(a)NGS分析显示NGS reads和Her2 NanoComb浓度呈正相关。(b)将SKBR3细胞和MCF7细胞培养到相似的密度,进行NanoDeep分析。NGS分析表明不同细胞株中Her2和Her3表达水平差异性(c)SKBR3 细胞经过饥饿处理后用HRG-β1(调节生长因子同系物)对其进行处理后进行NanoDeep分析。
进一步扩大结合文库,除了原有的EGFR亲和物文库,又另外增加了三种anti-CD63, anti-α5β1 和anti-CD71 结合文库来进行NanoDeep测试,在SKBR3细胞中增加的三种抗体亲和物相应膜蛋白只有CD71不和参考蛋白Her2结合,NanoDeep测试结果显示只有anti-CD71未能与NanoComb结合,反过来用CD71做为参考蛋白进行NanoDeep得到相同结果(图.7)。这些结果共同证明NanoDeep可以使用扩展的结合文库。NanoDeep有潜力作为研究EGFR家族受体信号和细胞膜中高阶结合作用的关键工具。
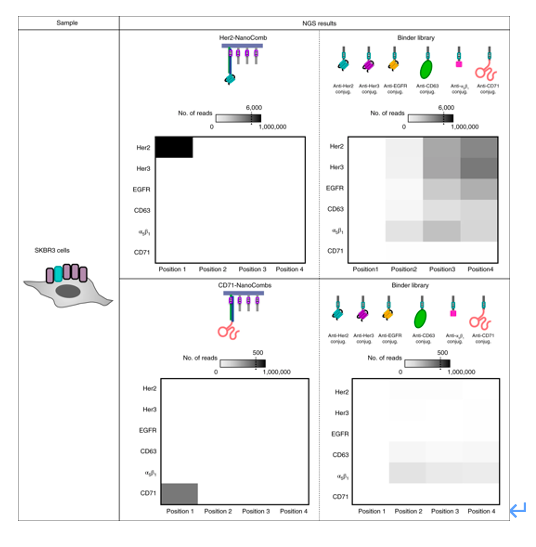
图.7 用扩展的结合文库进行NanoDeep检测
本文开发了一种分析细胞表面的纳米环境的方法,NanoDeep. 不同于超分辨率成像实现对单个纳米环境和允许实时成像而NanoDeep提供了整体细胞群的平均数据。利用DNA纳米技术制作的NanoComb可以提供高精度的膜蛋白的空间位置和组织信息。NanoDeep亲和文库类型的灵活性促进了亲和文库的进一步扩展。NanoDeep在不需要显微镜条件下同时分析膜上多种蛋白空间分布具有很大潜力,为理解膜蛋白组装提供了重要工具。
Ambrosetti, E., Bernardinelli, G., Hoffecker, I. et al. A DNA-nanoassembly-based approach to map membrane protein nanoenvironments. Nat. Nanotechnol. (2020). https://doi.org/10.1038/s41565-020-00785-0

