SCIENCE ADVANCES丨编程PAM触角以进行有效的CRISPR-Cas9 DNA编辑
作者:郑婷婷
细菌CRISPR-Cas9核酸酶已被重新应用为基因组编辑工具。尽管工程指导RNA或Cas核酸酶已被证明可以提高CRISPR编辑的效率,但对CRISPR不可或缺的前间区序列邻近基序(protospacer-adjacent motif,PAM)的调节却鲜有研究。PAM将sgRNA/Cas9招募至靶点及其对Cas9核酸酶结构域的激活对于CRISPR编辑是必不可少的,研究表明PAM密度的变化会影响Cas9与靶标的结合。对PAM空间分布和密度影响的研究可能会成为对CRISPR编辑进行工程改造的替代策略,并有助于更好地理解复杂环境(例如染色质)中的基因编辑。该文开发了一个基于DNA折纸的平台可对PAM微环境进行编程,并在单分子水平解决亚分子分辨率下的性能。在这项工作中,作者在目标DNA附近设计了DNA折纸限定的PAM触角,提供了一个微环境来研究PAM的空间分布对目标DNA切割效率的影响,并研究了在存在/不存在PAM触角的情况下sgRNA/Cas9结合的动态过程,并对PAM触角进行编程以实现更有效的DNA编辑。
为了在体外建立基于CRISPR-Cas9的结合和靶标切割的PAM环境,该文使用二维矩形DNA折纸作为模板来特异性定位sgRNA / Cas9组件,将双链DNA(dsDNA)目标置于折纸的中心。为了排列PAM触角,从DNA折纸DNA的3'末端延伸8条短链,使其邻近目标DNA,与包含6个PAM位点的单链DNA杂交,形成大约12 nm x 11 nm的富含PAM的区域(Origami P)。在DNA折纸的背面修饰了四个生物素分子,以将其固定在含抗生物素蛋白涂层的载玻片表面。将空白折纸(Origami B)与单独的目标DNA组装作为对照。AFM成像显示Origami P中心存在一个PAM岛,半峰宽约为16 nm,与预期宽度一致。

图1. DNA折纸上PAM触角的结构设计。
为了探讨PAM触角如何影响Cas9绑定,作者分别监测Cy5标记的sgRNA/Cas9与位于Origami B和Origami P上的目标DNA的结合。为此,将3'端延伸的sgRNA与Cy5标记的短ssDNA链杂交。为避免切割,在这里使用催化惰性的Cas9(dCas9)。通过分析目标DNA位置的荧光强度随时间的变化来表征dCas9的结合动力学。Origami P上的结合发生率(94±1.8%)大大高于Origami B(68±3.5%)。在单个折纸上的结合分布显示,Origami P上的dCas9富集程度高于Origami B,多重结合概率更高(n > 1),这表明PAM触角可作为dCas9绑定热点。同时,AFM高度分析也表明了PAM触角的存在使dCas9结合效率提高了。
为了证实PAM触角是Cas9的绑定热点,该文进行了马尔可夫链模拟,以研究核酸酶在携带PAM触角的折纸上的运动。通过绑定和解离速率来建模Cas9和PAM触角之间的瞬态相互作用(无绑定为Blank模型,具有高绑定率和高解离速率为non-PAM触角模型,具有高绑定率和低解离速率为PAM触角模型)。计算每个空间单元中Cas9的出现次数,获得了一个热图,显示了Cas9出现在每个单元上的概率分布。热图显示,Cas9的空间分布已在PAM触角的影响下重新排列,而解离速率高的non-PAM触角对Cas9的分布没有明显影响。
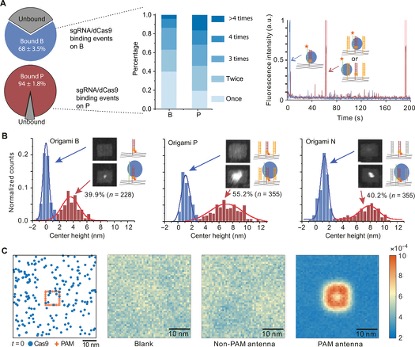
图2.PAM触角在矩形DNA折纸上创建了与Cas9结合的热点。
接下来,在单分子水平上探索PAM微环境对目标DNA裂解的影响,当在PAM近端标记Cy3时,大部分Cy3荧光迅速消失(图3B),表明Cy3标记的片段被切割和释放。相比之下,当在PAM远端标记Cy3时,释放过程明显较慢(图3C)。图3D分别监测Origami B,N和P上的目标DNA切割,以探索PAM触角的作用。带有PAM触角的Origami P上的目标DNA的裂解分数(87±4%)显着高于Origami B(77±1%)和Origami N(74±3%),这表明PAM触角的存在大大提高了目标DNA的裂解效率。
为了确定是否需要结构化PAM环境的形成来增强目标裂解,探索游离的PAM触角链对DNA目标裂解的影响(图3E)。结果表明在反应缓冲液中游离的PAM触角链会抑制目标DNA的切割,说明将PAM触角限制在结构化环境中对于提高切割效率是必要的。
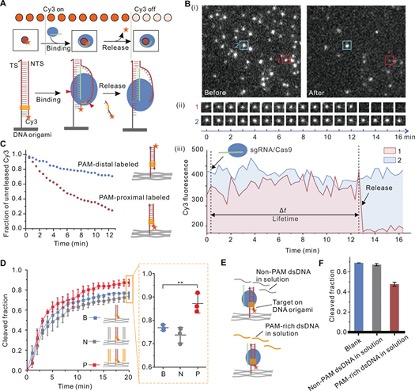
图3.PAM触角诱导增强目标DNA切割。
为了研究PAM触角提高目标切割效率的机制,作者在单分子水平上跟踪了dCas9在PAM触角内的运动。对单个dCas9的时间轨迹的分析表明,Origami P上的位移范围比Origami B上的位移范围大得多(图4B)。dCas9从其初始位置的位移的统计分析表明,dCas9在Origami B上200 ms内的最大位移范围在40 nm之内,而在Origami P上,位移范围增加到接近80 nm,这表明dCas9在PAM触角之间的局部扩散(图4A)。
为了进一步研究PAM微环境中Cas9的局部扩散机制如何导致增强的目标DNA裂解,作者使用随机游走模型对未结合的Cas9的空间分布进行了数值模拟(图4C)。在此模型中,Cas9在PAM内部执行短步长局部扩散,而在外部执行长步长自由扩散。图4D显示,在PAM触角内发现Cas9的可能性增加,并且在中心附近达到最大值。相反,在PAM触角内部和外部自由扩散的Cas9的模型预测未结合的Cas9的空间分布均匀,在中心没有显示增加的分布,这表明局部扩散模式对于增强目标结合至关重要。
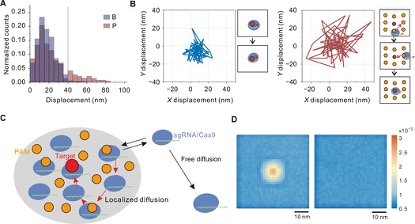
图4.局部扩散模式下目标搜索提高了目标结合率。
为了研究微环境中PAM触角的距离与密度如何影响目标DNA的切割,作者构建了两层PAM触角折纸(图5A,中)及富含PAM的折纸(图5A,右)。结果表明,有效距离内更高密度的PAM可以进一步提高靶标切割效率,因为更多的PAM可以增加募集可扩散至靶位的Cas9分子的能力。PAM密度与目标DNA切割效率之间呈正相关关系,表明可以通过排列PAM来调节微环境。
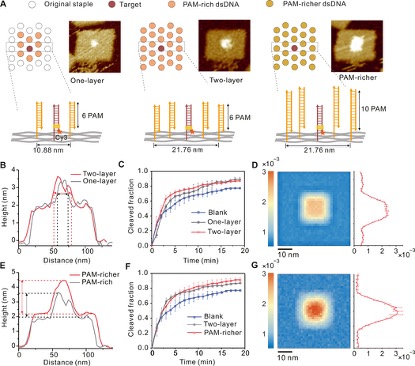
图5 PAM天线微环境调制程序设计。
在单分子水平上证实了PAM微环境的目标DNA切割增强作用之后,进一步验证是否可以对PAM微环境进行编程以进行更有效的批量基因编辑。作者设计了线性PAM触角,如图6A所示PAM邻近目标位点(T1),具有6 bp的非PAM序列间隔目标位点(T2),非PAM邻近目标位点(T3)。切割结果分析表明,PAM触角T1和T2邻近的目标DNA切割速度比T3快(图6B),且T1切割效率(97±1%)高于T3(88±5%)。比较T1和T2的切割动力学,发现二者对目标DNA的切割速度和最终裂解分数相近(图6B),表明目标DNA和PAM触角之间的短的非PAM间隔序列不妨碍Cas9的易位(图6A,中间)。Cas9跨非PAM区域的能力为更有效的基因编辑提供了更广泛的靶位点选择范围。
接下来,使用T1和T3进行结合观察,以研究与目标DNA相邻的线性PAM触角是否可以提高结合效率。为了便于识别,在DNA一端连接了链霉亲和素,在与sgRNA / dCas9孵育后,从AFM图像可见,sgRNA / dCas9在T1上结合位置有所变化,而dCas9在T3上占据特定位置(图6D)。该观察结果表明线性PAM触角可以募集Cas9分子,这些Cas9分子可以进一步转移到特定的靶位点,从而有助于提高切割效率。
为了探索sgRNA / Cas9浓度对有无PAM触角的目标DNA切割的影响,作者将T1和T3与不同比例的sgRNA / Cas9一起孵育,并通过凝胶分析定量了切割组分(图6E)。在每种浓度条件下,观察到的T1的切割效率均高于T3,这表明可以通过编程PAM触角使用更少的sgRNA / Cas9来实现基因编辑。且在低sgRNA / Cas9浓度下,T1与T3的切割组分之比显示出更高的切割增强率(图6E),从而有可能在CRISPR系统的递送效率受到限制时实现更有效的基因编辑。
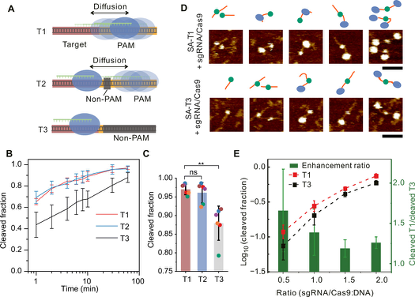
图6.线性相邻PAM触角可提高目标切割效率。
总结:
在这项工作中,作者在DNA折纸平台上与Cas9目标位点在空间上临近的位置设计了PAM触角,并研究了PAM触角对目标结合和切割的影响。在空间上锚定在目标DNA附近的PAM触角通过在目标周围募集更多Cas9分子来增强局部Cas9分布,进而提高目标切割效率。在体外和体内的许多生化反应中,结构化微环境都显示出至关重要的作用。然而,迄今为止,PAM微环境对CRISPR-Cas9基因编辑的影响一直被忽略。在这里,作者发现微调PAM微环境提供了一条提高基因编辑效率的新途径。通过对PAM触角进行编程,提高了结合和切割效率,为优化CRISPR-Cas9靶向位点提供了新的范例。(1)绑定到PAM触角的Cas9可以穿过短的非PAM间隔序列,从而允许结合并切割不直接位于PAM触角两侧的目标位点,选择范围更大,这表明对PAM触角进行编程具有改善基因组编辑的潜力效率和特异性。(2)在富含PAM的微环境中,可以以较低的sgRNA / Cas9浓度促进高效的基因编辑,这可以进一步减轻递送困难和对及细胞活性的影响,并提高CRISPR-Cas9基因组编辑系统的实用性。


