NAT COMMUN | 一种核酸存储的新方式
作者:张铭芷
使用DNA作为数据储存材料一直是科学家们热衷于探索的内容。在先前的研究中,通常使用人工合成的DNA以序列排列方式来存储数据,并通过自动化的二代测序进行数据读取。在William L. Hughes团队发表的工作中,使用了一种新颖的数据存储方式:使用DNA-PAINT技术,通过光斑阵列进行信息的读取。
在团队报道的数字核酸记忆(dNAM)中,主体部分是一个origami面板。其中staples至多包含两段序列:其一用于origami结构的形成,另一用于形成对接位点结合荧光探针。通过选择具有(1)或不具有(0)对接位点域的单链DNA的组合来编码数据。使用DNA-PAINT通过超分辨率显微镜监测荧光探针的结合,从而读取编码到面板中的信息。
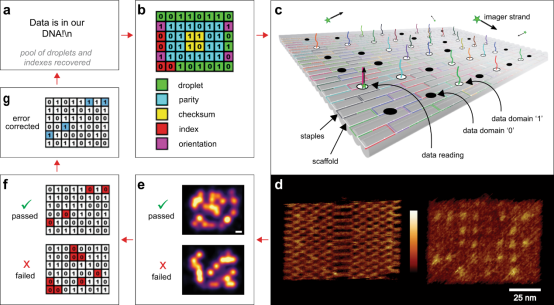
作者以存储及读取信息“Data is in our DNA!\n”为例展示了这一工作流程。首先,使用ASCII码对上述信息进行二进制转换,得到以01编码的数列。将数列以16位为一组分成10组,并对这10组信息使用喷泉码进行异或运算得到15组新的编码信息。同时,设置四位索引编码指示运算组合信息,并且设置四位方向信息。将新编码信息、索引信息和方向信息排布于一个6×8大小的矩阵中,填满矩阵的外边。
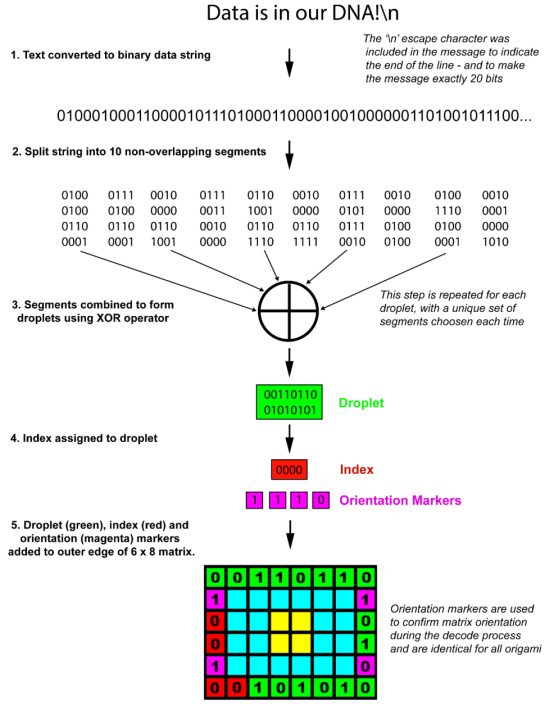
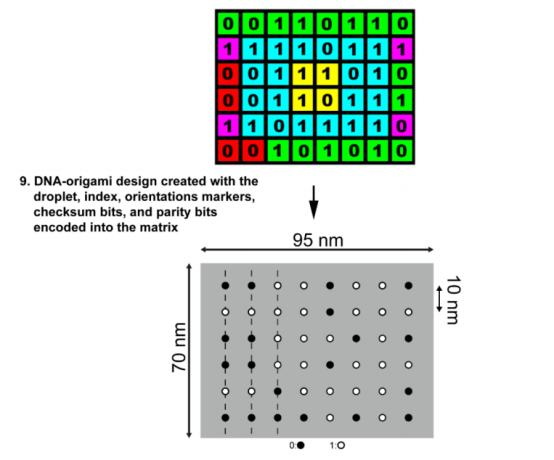
选择矩阵除边之外的内部区域为双级奇偶校验码,作为多层纠错使用,尽可能减少信息损失。编码完成后,将其设计成70 nm×95 nm的origami,其中每一检测点相距10 nm,矩阵中标记为1的点所用staple具有延伸端作为荧光探针识别区域,而标记为0的点所用staple不具有结合位点的延伸端。
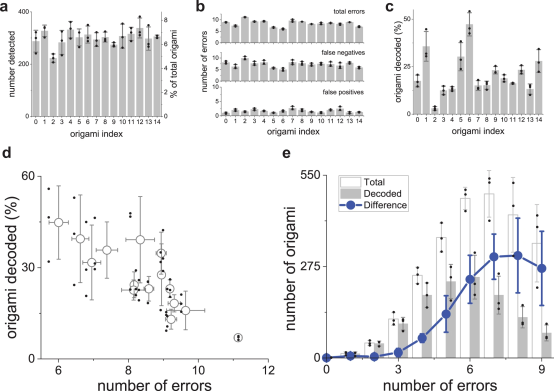
荧光探针结合后,使用TIRF对origami进行成像,通过多次采样和叠加得到较高分辨率的图样,进行origami上编码信息的读取。通过在所读取的01数列中提取编码信息和索引信息,借助从其他origami上提取的编码信息和索引信息可以通过逆运算得到原始编码信息。在这个过程中,荧光探针的结合情况和TIRF成像的质量会直接影响到原始信息的复原,因此需要先通过双级奇偶校验码进行可能的纠错程序。大致流程是通过校验码检查编码信息是否符合最初编码时的规范,若符合则接受;若不符合,将进行信息的随机突变,直至符合规范;若无法得到符合规范的突变,则弃去。这种使用喷泉算法叠加校验码的策略,很好地保证了信息存入和读取的真实性。并且在信息读出时,由于每一编码信息都包含了一种或多种原始信息,因此,只需要得到足够量的编码信息便可以将原始信息复原。

最后,作者评估了这种存储方法信息的保真度及解析出现的错误类型,为评估和优化未来dNAM设计的折纸性能提供了有用的指导。
该工作的结果证明了编写和读取寡核苷酸编码的数字信息的概念。由于DNA的持久性,dNAM在档案信息存储方面将具有长期的潜力。如果dNAM要与已建立的内存存储系统竞争,未来的工作还需要解决可扩展性问题。在该工作中,作者描述了少量数据的存储以说明dNAM的潜力。若扩展到更大的数据集,则需要在写入和读取速度方面进行实质性的工程改进。对于写入,限速步骤是选择寡核苷酸数据链。为了使dNAM使用实验室硬件达到相当于磁带(每秒数百MB)的写入速度,显著提高每条链的位数和样品转移速度或合成DNA寡核苷酸的速度非常必要。以具有竞争力的速度将信息写入DNA是整个DNA存储领域面临的严峻挑战,可能会随着DNA合成市场的增加而快速创新。


