NAT CHEM丨一种生物稳定的DNAzyme可以有效抑制细胞中基因的表达
作者:侯佳宁
本文作者是来自加州大学欧文分校药学院,化学院,分子生物学和生物化学学院的John C. Chaput和Robert C.Spitale。C. Chaput教授课题组一直以来致力于新类型XNA的开发,XNA Aptamer的功能改进与与靶标结合的结构分析,并将其用与疾病的诊断和治疗,以及对应的人工酶合成与改进。Robert C.Spitale教授课题组的主要方向是探寻RNA与疾病发病机制的关系,以及对RNA进行化学改性以增强其应用的能力。
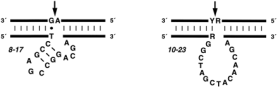
DNAzyme10–23,简称为10–23,是体外选择产生的Mg2+依赖性裂解RNA 的DNA酶。该酶包含一个15个核苷酸的(nt)催化结构域,该结构域的两侧为底物结合臂,底物结合臂长和序列具有特异性。与其他寡核苷酸治疗剂一样,RNA靶标可通过互补的沃森-克里克碱基配对识别。结合后,在预定的嘌呤-嘧啶连接处进行RNA切割,在GU之间的切割活性最高。裂解机理为从嘌呤(R)核苷酸对2'-羟基进行Mg2+辅助的去质子化,然后对相邻的磷酸二酯键进行亲核攻击,以产生具有2',3'-环状磷酸酯的上游裂解产物和下游裂解具有5'-羟基的产物。
多年来,研究者已经以各种方式对10-23进行了化学修饰,以提高体内和细胞内的功效。包括硫代磷酸酯键,2'-甲氧基修饰 ,倒3'-3'核苷酸胸苷,亚磷酰胺键和锁定核酸(LNA)。这些修饰对酶催化活性的影响,取决于残基的位置和化学修饰的类型。大多数化学修饰在基于增加与RNA靶标的亲和力的。但是,这种策略不利于DNA酶的催化活性,因为为增强RNA结合而选择的修饰通常会导致产物抑制,而酶-产物复合物无法从催化后状态解离(限制了上图中的K3)。同时,10-23的生物学稳定性不佳,难以抵抗生理环境下核酸酶的切割,以及酶活性的降低,阻碍了其临床试验的步伐,因此,10-23需要新的分子设计以有效地裂解细胞系统中的mRNA转录物。
整体设计:
作者报道了经典的10-23 DNA酶的重新设计版本X10-23,该酶介导了哺乳动物细胞中持久的基因沉默活性,同时还抵抗核酸酶的消化。研究者对DNA骨架上催化区域的每个位置进行结构突变,最终这些结构突变在模拟的生理条件下促进了增强的催化活性,X10-23具有DNA,2'-氟阿拉伯糖核酸和α- 1苏呋喃糖基核酸骨架结构,可在增强的生物稳定性与RNA杂交和二价金属离子配位作用之间取得平衡。在培养的哺乳动物细胞中,X10-23通过有效降解外源和内源信使RNA转录本来促进持久性基因沉默。这些结果表明,X10-23的活性和稳定性得到了提高,为其进入临床提供了可能。
结果与讨论:
1. 底物结合域的优化
作者首先用2'-氟阿拉伯糖核酸(FANA,图1b)替换了底物识别域结合臂中的所有DNA残基。先前的杂交研究表明,与相同的DNA-RNA双链体相比,FANA-RNA双链体每碱基对的稳定性通常高约1.2℃ 。F10-23的假一级速率常数几乎是未修饰亲本酶的两倍。为了提供RNA裂解活性的进一步证据,作者使用F10-23对核糖体修饰蛋白rimK的103 nt片段进行切割,该片段通过T7 RNA聚合酶的体外转录生成为未标记的RNA转录物。通过变性PAGE验证了其切割效果,与较短的RNA底物的观察结果一致,在所有测试的情况下,F10-23均比亲本10-23酶快。
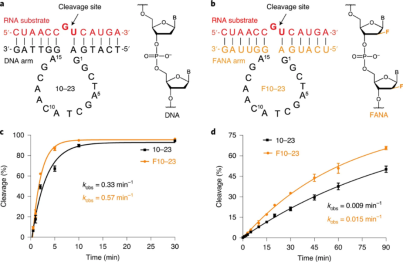
图1:a:左:10–23(黑色)与RNA底物(红色)复合。右图:DNA的化学结构。b:左:与RNA底物(红色)复合的F10-23酶(橙色)。右图:FANA化学结构。c,d:由10-23和F10-23进行RNA切割的稳态前动力学分析。反应在含有10 mM MgCl2(c)或1 mM MgCl2(d)和150 mM NaCl的缓冲液中于24℃(pH 7.5)进行,缓冲液含0.5 μM底物和2.5 μM酶。数据点指示底物裂解的百分比。
2. 催化区域的优化
接下来,作者对10-23的催化区域进行化学修饰以提高活性。作者用相应的FANA核苷酸系统地替换了催化核心中的每个DNA残基。在含有1 mM MgCl2的生理缓冲液中,测定了15种单点突变酶的完整组的RNA裂解活性。结果表明,残基G2和T8高度耐受FANA残基的取代,因为它们相对于亲本酶保持约80%的活性。该结果与先前的报告一致,该报告显示G2和T8接受由2'-5'磷酸二酯键引起的构象畸变。在其余的位置中,对T4和A9职位的替代表现出中等活性(50-60%),而C9,G14和A15职位的活性较低(20-30%)。同时带有G2和U8 FANA突变的10-23设计版本比亲本酶的活性高出近50%,表明这两个取代具有协同活性。
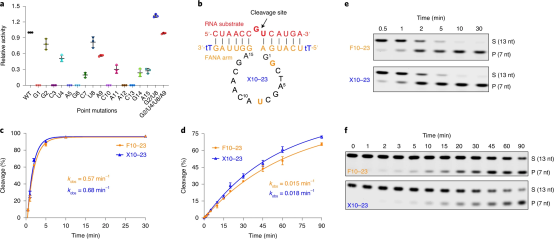
图2:a:由FANA取代的10-23催化中心区域的结构-活性图。数据点指示相对于野生型10-23 DNAzyme的相对底物裂解比例。b:X10-23酶与RNA底物复合。RNA,红色;DNA,黑色;FANA,橙色;TNA,蓝色。c,d:通过F10-23和X10-23进行RNA切割的稳态前动力学分析。反应在含有10 mM MgCl2(c)或1 mM MgCl2(d)和150 mM NaCl在24℃(pH 7.5)下使用0.5 μM底物和2.5 μM酶。点指示底物裂解的百分比。e,f :PAGE胶,显示在含有10 mM MgCl2(e)或1 mM MgCl2(f)的缓冲液中被修改过的10–23对RNA的切割作用。S,全长基板;P,5'裂解产物。
3. 整体改进后的催化活性和生物学稳定性测试
作者合成了一种新版本的酶,其中F10-23的催化核心经过修饰以包含G2和U8 FANA取代,并添加了非互补的α- 1-苏呋喃糖基胸苷(tT)残基在5'和3'末端的位置保护寡核苷酸免受核酸酶消化(图2b)。α-L-苏呋喃糖基核酸(TNA)是一种人工遗传聚合物,其中RNA中发现的天然五碳核糖已被非天然的四碳糖替代用于抵抗核酸酶切割。在稳态条件下用等摩尔浓度的底物和酶来评估10-23,F10-23和X10-23。动力学测量表明,F10-23和X10-23(图3a)比10–23快三倍,这与它们在稳态之前的活性一致。但是,当RNA底物的存在量超过酶的十倍摩尔过量时, F10-23和X10-23的活性比亲本酶高约50倍,这表明X10-23的催化前后状态与亲本10-23 DNA酶之间存在重要的结构差异。当亲本酶的结合臂延伸1 nt(7 + 7而不是6 + 6)时,没有观察到催化活性的差异(图3a)
除了有效的催化活性外,生物稳定性是RNA降解酶的细胞系统更关键的参数。作者分析了浓缩的人肝微粒体和DMEM中50%的人血清中10-23,F10-23和X10-23的稳定性。此外,还评估了其对蛇毒磷酸二酯酶(SVPDE)的抵抗力,蛇毒磷酸二酯酶是一种具有较强3'-核酸外切酶活性的攻击性酶,通常用于评估寡核苷酸治疗剂的稳定性。结果表明,X10-23在所有测试条件下均显示出显着增强的生物稳定性,证实了TNA作为用于保护5'和3'末端免于核酸酶消化的封端剂的实用性。
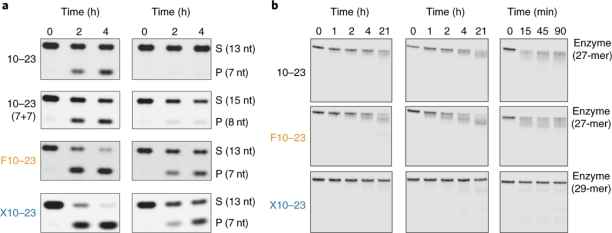
图3:a:PAGE胶,显示在稳态和多倍条件下的RNA裂解活性。RNA裂解反应在含有50 mM Tris-HCl(pH 7.5),1 mM MgCl2和150 mM NaCl的缓冲液中于24℃进行,带有0.5 μM底物和0.5 μM(稳态)(左)或50 nM(多周转)(右)酶。b:通过变性PAGE评估的时间依赖性生物稳定性测定。在2 ml 的人肝微粒体(左),50%的人血清(中)或10 mU/ml的SVPDE(右)的存在下,在含有1 μM酶的DMEM中评估RNA裂解酶在37℃下的生物学活性。
4. 与其他化学修饰版本的比较
作者合成了OME10-23和LNA10-23,OME10-23是10-23的类似物,其中16个DNA残基被2'- O-甲基核糖核苷酸(OME)取代(十个在底物结合臂中,六个在催化核心中),而LNA10-23是一个10–23类似物,其中每个结合臂中的三个末端DNA残基均被LNA取代。
动力学测量表明,在所有测试条件下,LNA10-23的速度明显快于OME10-23的速度。在底物和酶等摩尔浓度存在的稳态条件下,LNA10-23的活性优于OME10-23。(图4c)。但是,动力学曲线在多倍条件下有显着变化,其中LNA10-23显示出明显的产物抑制迹象(图4c)。因此,即使在单倍条件下LNA10–23比X10–23酶快,由于其在体外应用的特点,X10–23是在细胞应用的场景下效果更佳(图4d)
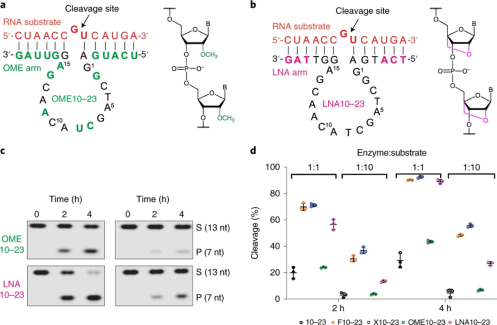
图4:a,b: OME10–23(a)和LNA10–23(b)与RNA底物复合。RNA,红色;DNA,黑色;OME,绿色;LNA,洋红色。c: 代表性的PAGE凝胶显示在稳态和多周转条件下的RNA切割活性。RNA裂解反应在含有50 mM Tris-HCl(pH 7.5),1 mM MgCl2和150 mM NaCl的缓冲液中于24℃进行,带有0.5 μM底物和0.5 μM(稳态)(左)或50 nM(右)酶。
d,不同化学取代的10-23活性的结构-活性图。数据点指示底物裂解的百分比。
5. 应用:
(1)细胞内绿色荧光蛋白的还原
作者使用绿色荧光蛋白(GFP)作为基因沉默活性的光学报告基因,研究了哺乳动物培养细胞中X10-23的活性。我们在存在和不存在两种X10-23试剂的情况下测量了GFP的表达,这些试剂被设计为靶向GFP mRNA转录物的编码(内部)和3'非翻译区(3'-UTR)中的GU二核苷酸(图5a)。使用HEK293T(HEK)细胞进行分析,然后将HEK293T(HEK)细胞转染由CMV(人类巨细胞病毒)启动子驱动的GFP表达质粒。转染后24小时孵育后收集的荧光图像显示,与GFP质粒和X10-23试剂同时转染内部位点或3'-UTR位点或两个位点同时转染的细胞,GFP信号丢失与仅用GFP质粒转染的细胞相比(图5b)。定量逆转录PCR(qRT-PCR)测量证实GFP信号的丢失是由于GFP的mRNA模板拷贝数下降所致(图5c),这表明细胞中蛋白质和mRNA水平均降低。
(同时也对于CMV启动子强度的影响进行了扣除实验)
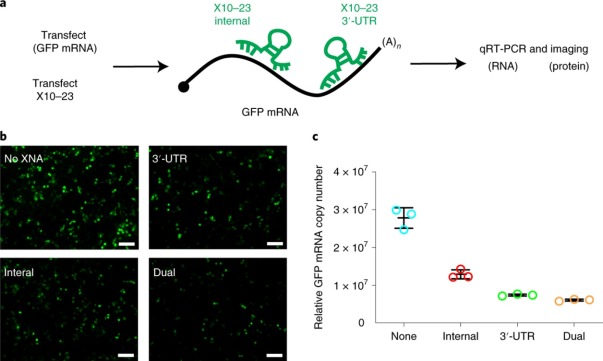
图5:a: 用于细胞内抑制GFP的X10-23分子的示意图。HEK293细胞被靶向GFP mRNA转录物中两个不同位点的X10-23分子共转染。通过GFP荧光细胞成像和qRT-PCR测量基因沉默活性的水平。b: 在转染后24小时收获细胞之前收集的GFP荧光细胞图像。比例尺: 100 µm。c: qRT-PCR分析转染后24小时使用GFP特异性和GAPDH(3-磷酸甘油醛脱氢酶)上样对照引物分离的无DNA的总RNA。
(2)内源性KRAS的细胞内还原
作者以KRAS细胞靶标,设计,合成和测试了两种针对第一个外显子和3'-UTR的X10-23试剂(图6a)在宫颈癌(HeLa)和乳腺癌(MDA-MB-231)细胞系中的内源性KRAS。HeLa和MDA-MB-231细胞在有或没有4μgX10-23的情况下进行了转染,转染后孵育48小时后,通过qRT-PCR定量了KRAS mRNA水平(图6b)。相对于转染对照,两种细胞系均显示针对第一个外显子的X10-23试剂的mRNA拷贝数减少了> 65%(图6c,d)。靶向3'-UTR的X10-23试剂效果稍差,KRAS mRNA拷贝数降低了约35-45%(图6c,d)。两种X10–23试剂之间的RNA裂解活性差异与合成寡核苷酸的体外活性一致并且可能反映了这两种试剂结合能的序列特异性差异。表明X10-23可用于敲低人类细胞中致病蛋白的表达。
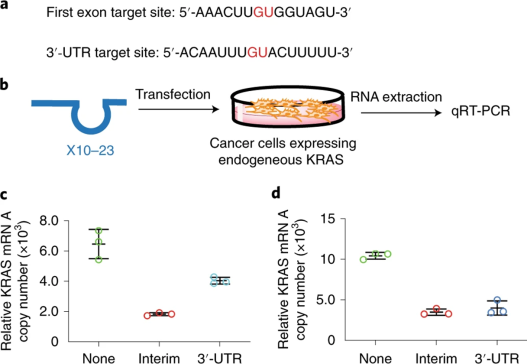
图6:a: X10–23目标位点位于KRAS的第一个外显子和3'-UTR区域。裂解GU为红色。b,用于抑制癌细胞表达的内源性KRAS的X10-23分子的示意图。宫颈癌细胞(HeLa)和乳腺癌细胞(MDA-MB-231)分别用单独的X10-23单个X10-23或有转染载体但没有X10-23,并且通过qRT-PCR定量分析了KRAS mRNA的拷贝数。c,d: 从HeLa细胞(c)和MDA-MB-231细胞(d)中提取的无DNA总RNA的qRT-PCR分析。转染后48小时,使用KRAS特异性和GAPDH上样对照引物。
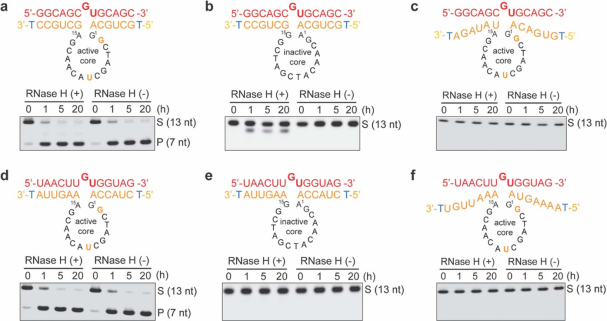
补充图1:代表性的凝胶显示在有和无RNase H的情况下对GFP的内部片段(a-c)和KRAS RNA的第一个外显子片段(d-f)具有RNA切割活性。RNA(红色),DNA(黑色),FANA(橙色)和TNA(蓝色)。(a,d)具有活性催化核的X10-23。(b,e) X10-23,具有惰性催化核。(c,f)具有不与RNA靶杂交的活性催化核心的X10-23。所有测定均在37℃(pH 7.5)的含0.5 mM MgCl2和150 mM NaCl的缓冲液中进行,缓冲液含1 μM底物和1 μM酶。核酸酶反应包括0.1 U/μL的RNaseH。S:全长底物,P:5'裂解产物。分子量标记显示在凝胶的右侧。
总结:
DNAzyme具有序列特异性切割RNA,易于通过固相化学合成获得,易于定向进化,且可对任何感兴趣的RNA靶向切除的特点,但是临床试验中酶活性的降低极大阻碍了其向临床的转化。当前的研究旨在通过扩大用于构建核酸酶的核酸类似物来缩小DNA酶和基于蛋白质的基因沉默工具之间的差距。
作者利用XNA这种具有新型糖磷酸盐骨架的人工遗传聚合物,对传统DNAzyme的核酸骨架结构中的适当定位进行改进,实现生理条件下增强的RNA裂解活性,并同时保护分子免受核酸酶消化。
1. 作者系统地研究了经典10-23的底物结合臂和催化结构域,发现了一种高效且生物稳定的变体,其中包含三类不同的核酸分子(DNA,FANA和TNA)。
2. 相对于亲本酶,X10-23在模拟生理条件下的催化效率提高了约50倍,骨架结构的生物稳定性提高了100倍以上,同时作者证明了X10-23不依赖RNase H作为RNA降解的机制。
3. 在培养的哺乳动物细胞中, X10-23靶向的mRNA和蛋白质丰度降低60%以上,在用转录抑制剂处理时,其进一步增强。对于靶向人类癌细胞系中内源性KRAS表达的X10–23,观察到了相似作用,这表明X10–23可能对难成靶蛋白质的有一定的应用前景,来推动精密医学领域的发展。
Wang, Y., Nguyen, K., Spitale, R.C. et al. A biologically stable DNAzyme that efficiently silences gene expression in cells. Nat. Chem. 13, 319–326 (2021). https://doi.org/10.1038/s41557-021-00645-x

